4.2. Replicación del DNA en eucariontes.
El mecanismo para la síntesis del DNA en eucariontes probablemente es similar al proceso en procariontes.
Es similar a la de los procariontes, es decir,
semiconservativa y bidireccional. Existe una hebra conductora que sintetiza de manera continua y la retardada de forma discontinua con fragmentos de Okazaki.
La enzima que lleva a cabo la replicación del ADN es la
ADN polimerasa, esta enzima tiene unos requerimientos específicos para trabajar, que le imponen restricciones:
1. Sólo añade nucleótidos en la dirección
5’ 3’.
2. Necesita para poder empezar a copiar y unir nucleótidos un
molde de ADN.
3. Necesita un pequeño trocito de ARN al cual unir los nucleótidos, ya que ella no puede empezar a unir los nucleótidos sin tener una pequeña cadena ya formada.
4. Utiliza nucleótidos trifosfato.
Dadas las necesidades de esta enzima y sabiendo que la molécula de ADN está formada por dos hebras antiparalelas, se plantea un problema en la cadena de ADN que va en la dirección 5’
3’, porque aquí la enzima estaría trabajando en la dirección contraria, y sin embargo, se observa que las dos hebras se van replicando.
La solución al dilema la dio el desabrimiento, en 1968, por
Okazaki de unos fragmentos constituidos por unos 50 nucleótidos de ARN y entre 1.000 y 2.000 nucleótidos de ADN, denominados fragmentos de Okazaki. Así se podía explicar como se copia esta cadena, siendo de forma discontinua en la dirección 5’ 3’ y la otra cadena de forma continua. También quedaba solucionado el problema de que la enzima necesitara una cadena de ARN a la cual unir los nucleótidos.
Existe una secuencia de nucleótidos en el ADN llamada origen de replicación que actúa como señal de iniciación.
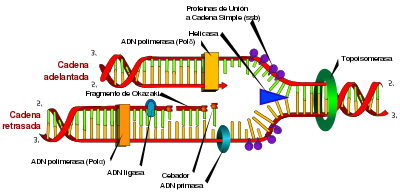
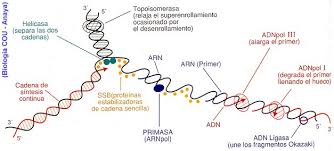
Las cadenas de ADN están unidas por puentes de hidrógeno, que debemos romper para facilitar la separación de las cadenas para ser copiadas, esta separación la lleva a cabo las enzimas helicasas.
Como el desenrollamiento de la doble hélice da lugar a superenrollamientos en el resto de la molécula, capaces de detener el proceso, se hace preciso la presencia de las enzimas topoisomerasas que eliminen las tensiones en la fibra.
A continuación, para evitar que las dos hebras vuelvan a reunirse y formar los puentes de hidrógeno se colocan unas proteínas llamadas SSB
El proceso es bidireccional, es decir, hay una helicasa trabajando en un sentido y otra trabajando en sentido opuesto. Se forman pues las llamadas burbujas u ojos de replicación.
Como ninguna ADN-polimerasa puede actuar sin cebador, interviene primero una ARN polimerasa (primasa) que si lo puede hacer, sintetiza un corto fragmento de ARN de unos 10 nucleótidos denominado primer que actúa como cebador.
Después interviene la ADN polimerasa III, que a partir de este cebador comienza a sintetizar en dirección 5’ 3’ una hebra de ADN partir de nucleótidos trifosfato. La energía necesaria para el proceso es aportada por los propios nucleótidos que pierden dos de sus fósforos. Esta nueva hebra se sintetiza en el sentido que se abre la horquilla de replicación, es de crecimiento continuo y se denomina hebra conductora.
Sobre la otra hebra (hebra discontinua o retardada) la ARN polimerasa sintetiza unos 40 nucleótidos de ARN en un punto que dista unos 1.000 nucleótidos de la señal de iniciación. A partir de ellos la ADN polimerasa III sintetiza unos 1.000 nucleótidos de ADN, formándose un fragmento de Okazaki. Este proceso se va repitiendo a medida que se van separando las dos hebras patrón.
A continuación interviene la ADN polimerasa I, que, primero, gracias a su función exonucleasa, retira los segmentos de ARN, y que luego, gracias a su función polimerasa, rellena los huecos con nucelótidos de ADN.
Finalmente la ADN ligasa unirá los dos extremos, tanto en la cadena continua como los sucesivos fragmentos de Okazaki que se van formando en la cadena discontinua.
Cuando se observa como se produce la replicación se ve que la cadena continua va más rápida que la discontinua, esto es debido a que cada vez que se forma un fragmento de Okazaki hay que realizar todo este proceso como si se comenzara la síntesis de nuevo, formando el primer y el resto de pasos, sin embargo, en la continua solo se realizará una vez.
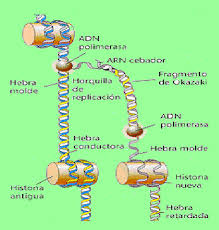

El ADN de los eucariontes está fuertemente asociado a los octámeros de histonas, en forma de nucleosomas, por lo que además de replicarse el ADN, deben duplicarse también las histonas. Al parecer, tanto los nuevos nucleosomas como los antiguos se reparten de manera aleatoria entre las dos nuevas hebras hijas: en la retardada y en la conductora.
La longitud del ADN de un cromosoma eucariótico es mucho mayor que el ADN bacteriano, de ahí que no haya un único origen de replicación. Para que el proceso sea más rápido, existen numerosas burbujas de replicación a lo largo de cada cromosoma.
Terminación de la replicación: síntesis de los telómeros
El final de la replicación en eucariotas tiene que resolver el problema del acortamiento de los cromosomas. Para ello ha desarrollado la estructura de los telómeros y telosomas, y la actuación de una nueva enzima emparentada con las retrotranscriptasas: la telomerasa.